Die Systeme des Multisuchsystems
Alle Systeme sind ausschliesslich
JavaScript-Anwendungen in
Verbindung mit Webformularen. Die gesamte Technik ist in der
Publikation Informationen
erfassen und bearbeiten ausführlich
erläutert.
Ein wesentlicher Teil dieser Technik und die
Basis für
alle im Multisuchsystem enthaltenen Systeme ist die
Datenübernahme, die durch Copy und Paste geschieht. Hierbei
werden
im allgemeinen vollständige Webseiten kopiert und in ein
Formularfeld eingefügt und anschliessend die auf den Webseiten
enthaltenen Informationen wie Zeitschriftentitel oder Aufsatzzitate
übernommen und bearbeitet. Schematisch
lässt sich
das Verfahren so darstellen:
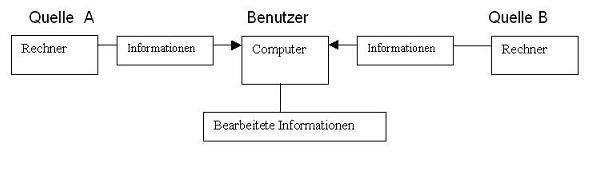
Mit Ihrem Computer übernehmen Sie direkt die Informationen der
Quellen A und B, unabhängig von einem Zwischen-Anbieter. Diese
werden in Ihrem Computer gespeichert oder nur temporär
übernommen, wo sie anschliessend nach verschiedenen Kriterien
bearbeitet und durchsucht werden können.
Die Quelle A können hierbei die von verschiedenen Anbietern
(z.B.
der EZB) angezeigten Zeitschriftenlisten, die Quelle B die von
Datenbanken angezeigten Suchergebnisse sein. Diese beiden Quellen
werden durch ein auf Webformularen basierendem JavaScript-Programm
aufgeschlüsselt und deren einzelne Elemente miteinander
verknüpft. Vor allem werden die Zeitschriftentitel aus den
Zitaten
erfaßt und mit den Titeln der übernommenen
Zeitschriftenlisten verknüpft. So werden die
zusätzlichen
Informationen gewonnen, die in Datenbankergebnissen nicht oder nur
unzureichend gegeben sind, z.B. die Verfügbarkeit von
Volltexten
und deren Zugänglichkeit. So können auch umfangreiche
Literaturverwaltungssysteme leicht erstellt und diese wiederum mit der
Zeitschriftenverwaltung verbunden werden, so daß ganz
neuartige
Systeme wie z.B. Contents-Linking II entstehen kann.
Datenbanken und Einarbeitungssysteme
ASLA. ASEZA, Contents-LInking I, II und
Contents-LinkingB
enthalten umfangreiche erweiterbare Datenbanken von
Zeitschriftentiteln von 76000 bzw über 16000 Titel, je nachdem
ob
der grössere Gesamtbestand der EZB oder der Bestand der
Thomson
Reuters Journal List gewählt wird. Ein
Sonderfall ist Contents-Linking II, das ausser einer
Zeitschriftentiteldatenbank auch eine Aufsatzdatenbank von ca. 46000
Zitaten enthält, die ebenfalls erweiterbar ist.
Diese
Systeme können als abgeschlossene Systeme mit den aktuell
vorliegenden Datenbanken arbeiten. Sie können aber auch
mit neuen Daten ergänzt oder durch neue Daten ersetzt werden.
In
bezug auf die Zeitschriftendatenbanken sind dafür die
Einarbeitungssysteme einarb.htm
für ASLA, ASEZA, Contents-Linking
I und II und einarbB.htm
für Contents-LinkingB vorgesehen.
Für die Aufsatzdatenbank ist das Multiple
Linksystem zu verwenden,
mit dem aus wichtigen bibliographischen Datenbanken und mit
verschiedenen Formaten Zitate in grosser Anzahl (bis mehrere Tausend auf einmal!) übernommen
und eingespeichert werden können.
Dabei
kommt, wie auch bei den Einarbeitungssystemen, die ActiveX-Technik zur
Anwendung.
Das Multiple Linksystem kann ausserdem als
universeller
Linkresolver für eine temporäre Anwendung eingesetzt
werden,
wobei die eingefügten Zitate bearbeitet und mit
Verknüpfungen, die vor allem zum Volltext hinführen,
angezeigt werden. Diese Arbeitsweise entspricht ganz derjenigen der
früheren Linksysteme
des Multisuchsystems. Das Multiple Linksystem
ersetzt also alle früheren Linksysteme, die von mir in einer
Reihe von Publikationen dargestellt wurden:
Hehl,
H. Der Recherchedienst Ingenta und seine
Erweiterung
durch das Linksystem Ingenta-Link
In:
BIBLIOTHEK FORSCHUNG UND PRAXIS 2002,
Vol. 26, T. 2, S. 169-175
Hehl,
H. Das Linksystem
Math-Link
In:
BIBLIOTHEKSDIENST 2001, Vol. 35, T. 10,
S. 1344-1350 0006-1972
Hehl,
H. Medline-LINK. Ein Linksystem zur
Integration von Literatursu
In:
NFD INFORMATION 2000, Vol. 51, T. 4, S.
209-216
Hehl,
J. Linksysteme - Einfache und effektive
Verfahren mittels Webformularen
In:
INFORMATION
WISSENSCHAFT UND PRAXIS 2005, Vol. 56,
Nr. 5/6, S.
303-308 1434-4653
Hehl,
J. SFX und die Linksysteme im
Multisuchsystem E-Connect. Ein Vergleich
In:
BIBLIOTHEKSDIENST 2005, Vol. 39, Nr. 7,
S. 932-945 0006-1972
Hehl,
H. Die Linksysteme des Multisuchsystems
E-Connect.
In:
BIBLIOTHEKSDIENST 2003, Vol. 37, T. 6,
S. 774-790 0006-1972
Hehl, H Die ASEZA-Datenbank: ein
neues Suchsystems für elektronische Zeitschriften und
Aufsätze
in: ABI - Technik 2012, 32, 14 - 25
Das
Link-System WebSPIRS-Link
in: B.I.T.
Link-Systeme
mittels JavaScript zur Integration von Literatursuche und
Literaturbeschaffung,
in: B.I.T.online, Heft 1/2000
Übersicht über die Systeme und Dateien des
Multisuchsystems
Die einzelnen Systeme und ihre Verwendung
- ASLA
Citation Linker für ein
einzelnes Zitat,
für die gezielte Suche nach einem Zeitschriftentitel, aber
auch für die nichtgezielte sachliche
Suche nach
Schlagwörtern
- ASEZA
Suche nach Zeitschriften über
Zeitschriftentitel,
Fachwörter und Schlagwörter
- Contents-Linking
I wie ASEZA
- Contents-Linking
II wie Contents-Linking I, verbunden mit Aufsatzdatenbank,
Literaturverwaltung
- Contents-LinkinB
vereinfachtes Contents-Linking I
- Multiples
Linksystem temporäres Linksystem für Zitate
oder Einarbeitungssystem
für Contents-Linking II
Der Aufbau der Systeme
- Contents-Linking I
contents4.html
Startformular
suchcont3a.htm Schlagwortauswahl
contents4.js Javascript-Datei
such1.htm Zeitschriftendatenbank: G_G.txt (z.Zt
(29.3.04) ca. 76000 Titel), ca. 540 Schlagwörter
- Contents-Linking II
contents3.html
Startformular
suchcont1a.htm Zeitschriftendatenbank: G_G.txt+Pubmed (ca. 101000
Titel) ScidatZZ, Schlagwörter
contents3.js Javascript-Datei
suchcont1b.htm Aufsatzdatenbank (ca. 47000 Zitate): scidatA.txt
suchcont3.htm Schlagwortsuche
Beschreibung
in: ContentsNA.PDF
- Contents-LinkingB
contentB.htm
Startformular: javascript, Zeitschriften (G_G.txt ca. 76000 Titel),
Schlagwörter
Beschreibung:
entdcontent.PDF
4. ASLA
asla.htm Startseite
such3.htm Datenbank (G_G.txt)
suche3b.js Javascript-Datei
fenster.htm Zusatzfenster
Themen4.htm Themendatei
aslaE.htm
Erläuterung
helpmult.htm Suchhilfe
Arbeitsweise von ASLA
Über
den Button Suche starten wird die Funktion starte()
aufgerufen. In dieser werden die Werte aus dem Formular
gelesen und mit diesen Werten die Adresse
such3.htm?au=&&b=&&c=j
chrom&&cb=&&th=&&BI=AAA&&d=&&v=&&n=&&pg=&&pa=0
generiert, wobei such3.htm
die Datenbank enthält. Diese Adresse wird
angewählt.
such3.htm ist mit der Javascript-Datei suche3b.js verbunden. Beim Laden
wird darin die Funktion starte()
aufgerufen. Darin werden die in der Adresse enthaltenen Werte
ausgelesen. Mit den für die Zeitschrift entnommenen Werten
werden
in der Datenbank die Titel ermittelt, die diesen Werten entsprechen.
Die Titel werden mit den Verknüpfungen ausgeschrieben, wobei
die
aus dem Formular und der Datenbank entnommenen Werte
berücksichtigt werden.
Themen
und Fachwörter
werden über das Zusatzfenster Themen4.htm ausgewählt,
das die
Themenliste enthält. Themen werden durch Anklicken in das Feld
des
Formulars eingetragen. Bei Fachwörterauswahl wird die
Datenbank such3.htm gestartet und die Funktion starte()
aufgerufen. Aus der Adresse wird der Wert fw=... gelesen
und dementsprechend die Function fachww()
aufgerufen. In dieser werden alle Fachwörter des betreffenden
Themas aus allen Titeln der Datenbank ermittelt, mit einer Datei von
Nichfachwörtern verglichen und angezeigt. Beim anklicken
Eintragung in das oberste Feld des Formulars.
Weiter
Beschreibung in: ASLA und Citation
Linker.PDF
5. ASEZA (ist identisch
mit Contents-Linking I ausser der Startseite)
aseza.html
Startseite-Formular
suchcont3a.htm Schlagwortauswahl wie in Contents-Linking !
such1.htm Datenbank wie in Contents-Linking I
contents4.js
Javascript-Datei wie in Contents-Linking I
Beschreibung: suchAsezaE.htm
6.
Einarbeitungssysteme für
Zeitschriften
- einarb.htm
für ASLA, ASEZA,
Contents-Linking I und II
- einarbB.htm für
Contents-LnkingB
Einarbeitung I einarb.htm
einarb.htm übergeordnete Datei
einarb2.htm Formulardatei
such1a.htm Datenbank (G_G.txt)
such1g.htm Serien-Titel, gleichlautende Titel,Titel mit
Abkürzungen
Arbeitsweise: 3 Stufen-Ablauf:
Start1: function start_b()
Datenbank ISI_G.txt=such1a wird ausgelesen,
eingefügte Liste verändert,
einzelne Titel mit den Titeln in such1g verglichen,dann mit Titeln in
Datenbank
verglichen, Ergebnisse in Feldern area6 (Serien u. gleichlautende),
area5
(nichtgefundene), area1a, area1b, area1c (gefunden)zwischengelagert
Start2: function start()
Datenbank wird ausgelesen, ebenso die
gefundenen eingefügten Titel. In einer
For-Schleife werden die Titel der Datenbank mit deneingefügten
Titeln
verglichen. Identische Titel erhalten ggf. die ergänzten oder
berichtigten
Angaben zu Schlagwörtern und Verfügbarkeit.
Ergebnis in area2, area3, area4 gespeichert.
Start3: function start2()
Titel in area2, area3. area4, evtl. auch
nichtgefundene in area5 werden
ausgelesen, das Ganze etwas verändert und sortiert,als Ta5
ausgeschrieben.
Anschliessend Lesen der Dateien:
suchcont1a.txt einleitender Text für
suchcont1a.htm
med.txt PubMed-Zeitschriften
scidatZZ.txt in Aufsatzdatenbank suchcont1b.htm enthaltene Zeitschriften
such1.txt einleitener Text für such1.htm
Themen.txt Schlagwortliste
contentB.txt Textdatei ohne Datenbank für contentB.htm
ausgeschrieben werden:
G_G.txt Bestandsliste
such1.htm Bestand (G_G.txt) , Themen für
Contents-Linking I
such1a.htm Bestand (G_G.txt)
) für Einarbeitungssystem
suchcont1a.htm Bestand (G_G.txt + PubMed), ScidatZZ, Themen
contZSS.txt Bestand G_G.txt + PubMed in suchcont1a
enthalten)
Einarbeitung II einarbB.htm
einarbB.htm übergeordnete Datei
einarb2b.htm Formulardatei
g_g.htm Datenbank (G_G.txt )
such1g.htm (Serien-Titel, gleichlautende Titel,Titel mit
Abkürzungen)
Gelesen werden: g_g.htm (G_G.txt aus Formular), Themen.txt,
ContentB.txt)
Geschrieben werden: G_G.txt, contentB.htm, g_g.htm
Einarbeitung für ASLA: Zugang
unten Übernahme
eines neuen Bestandes aus der Datei G_G.txt ,
Function such3sp()
such3.txt, G_G.txt,
Fachwörter2.txt, Themen.txt werden gelesen,
such3.htm wird ausgeschrieben
7.
Multiples Linksystem (Linkresolver und Einarbeitungssystem für
Zitate)
AusgabeNa.htm
Button Starten
und anzeigen
function
starte() Zitate bearbeitet und in Feld dat
abgelegt, anschliessend:
function anz() Zitate mit
Verknüpfungen
ausgeschrieben
Speichern
in Contents-Linking
function
hinzu2()
Zitate aus Feld dat auslesen
gelesen werden die Dateien: suchcont1b.txt (einleitender Text
für
suchcont1b.htm), scidatA.txt (Aufsatzdatei)
geschrieben werden: scidatAA.txt, suchcont1b.htm
anschliessend:
function spei()
gelesen
wird: scidatAA.txt (Aufsatzdatenbank Kopie)
geschrieben wird:
scidatA.txt
Zeitschriften
anzeigen und aktualisieren
gelesen
wird: scidatAA.txt, scidatZ.txt
geschrieben wird: scidatZZ.txt
anschliessend:
gelesen
werden: suchcont1a.txt, contZSS.txt, scidatZZ.txt, Themen.txt
geschrieben werden: suchcont1a.htm, scidatZ.txt
8.
Eingabeformular für neue Formate: Eingabeformular.htm
Zitate übernehmen:
function anderes()
fensterNe2a.htm Schema
für Datenübernahme wird
gestartet
Daten
übernehmen:
Suchstring
wird ausgeschrieben (kann in die Datei AusgabeNa.htm für ein
neues Format eingefügt werden)
Die Zeitschftendatenbanken in
ASLA, ASEZA und
Contents-Linking
ASLA:
such3.htm (G_G.txt +Themen)
ASEZA:
such1.htm (G_G.txt +Themen)
Contents-Linking
I: wie ASEZA
Contents-Linking
II:
suchcont1a.htm (G_G.txt + med.txt + scidatZZ.txt + Themen)
Contents-LinkingB: contentB.htm (G_G.txt +Themen)
Die
Textdateien der Datenbanken
G-File.txt
Gesamtdatei EZB mit und ohne ISSN
G-FileA.txt
Ersatzdatei
GesamtISSNPlus.txt Gesamtdatei von Titeln mit ISSN
(haupsächlich ISI-Titel)
G_G.txt aktualisierte Gesamtdatei mit
Beständen mit und ohne ISSN ggf. mit ISI_G.txt
austauschen!
ISI_G.txt aktualisierte ISI-Titel mit
Beständen ggf. mit G_G.txt austauschen!
pubmed.txt originale PubMed-Titel
med.txt aktualisierte
PubMed-Titel (ca. 24000) mit Beständen für
Contents-Linking II
contZSS.txt G_G.txt und PubMed-Titel
scidatZZ.txt in der
Aufsatzdatenbank von Contents-Linking II enthaltene Titel (Kopie)
scidatZ.txt in der Aufsatzdatenbank von
Contents-Linking II enthaltene Titel
isijournals originale ISI-Journals
isijournals2 bearbeitete ISI-Journals
gesamtezb.txt originale
EZB-Titel kopiert
gesamtezb2.txt EZB-Titel
bearbeitet
scidatA.txt Aufsatzdatenbank
scidatAA.txt
Aufsatzdatenbank Kopie
Dateien mit Bestands- und Themenangaben: G_G.txt,
ISI_G.txt,
med.txt,
contZSS.txt
Die
wichtigsten Bearbeitungssysteme
BearbEZB3.htm
gesamtezb2.txt mit GesamtISSNPlus.txt
vergleichen,
Basisdatei G-File.txt entsteht = EZB-Titel mit ISSN
ergänzt
BearbISI_G.htm
Bearbeitung der ISI-Titel
BearbISI_G2.htm ISI_G und med.txt
aktualisieren:
isijournals3.txt mit
G_G.txt vergleichen
und ergänzen
med.txt mit ISI_G.txt
vergleichen und
ergänzen
BearbG.htm
Bearbeitungen der aktualisierten Datei
G_G.txt
BearbG_G.htm Aktualisierungen der Bestandsdateien G_G.txt, such1.htm, such3.htm und suchcont1a.htm
Bearbscidat.htm Bearbeitung der Aufsatzdatei scidat.txt
Weitere
Informationen zu den Bearbeitungssystemen siehe Bestandsliste.PDF.
Alle
Einarbeitungs- und Bearbeitungssysteme arbeiten nur in einer lokal
installierten Version mit dem IE!
©
2014 Hans
Hehl,
Regensburg
